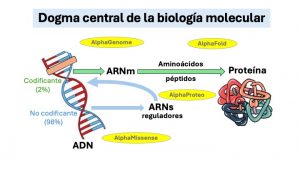
El dogma central de la biología molecular fue formulado por Francis Crick en 1958. La vida se sustenta en un flujo unidireccional de la información genética dentro de cada célula: el ADN se replica, se transcribe a ARN mensajero y éste de traduce en aminoácidos que conforman proteínas.
En el año 2010 nació la empresa DeepMind en Inglaterra, con el objetivo de desarrollar aplicaciones avanzadas de computación e inteligencia artificial (IA). En 2014 fue adquirida por Google. En el momento actual, Google DeepMind es una empresa biotecnológica de investigación en IA del grupo Alphabet (Google).
Los principales proyectos de Google DeepMind en biología computacional se centran en la comprensión detallada de los mecanismos de actuación y la función de los elementos del dogma central de la biología molecular. En otras palabras, son programas que descifran los mecanismos de la vida y, de ese modo, podrían permitir que podamos intervenir y modificarlos. De forma ideal, ayudarán a prevenir enfermedades y desarrollar nuevos tratamientos.
Los ‘4 Alphas’ de Google DeepMind
En los últimos 5 años, Google DeepMind ha puesto a punto cuatro grandes herramientas biotecnológicas: AlphaFold, AlphaMissense, AlphaGenome y AlphaProteo.
AlphaFold es un programa que fue presentado en 2018, pero su versión con mayor precisión es de 2020 (Jumper y cols. Nature 2021). Ha supuesto una revolución en la predicción de la estructura tridimensional de proteínas a partir de la secuencia de aminoácidos. Sus diseñadores fueron reconocidos con el premio Nobel de Química 2024.
AlphaMissense predice los efectos de mutaciones ‘missense’ -es decir, que producen cambios en aminoácido- para predecir si una variante genética puede ser dañina o benigna. Puede ser de gran utilidad para el diagnóstico genético rápido, por ejemplo identificando variantes que causan enfermedades raras (Cheng y cols. Science 2023).
AlphaGenome es un programa de IA que analiza secuencias largas del ADN (hasta 1 millón de bases) y predice cómo las mutaciones pueden afectar la regulación de los genes. De ese modo, ayuda a comprender distintos rasgos fisiológicos o enfermedades genéticas, la biología del desarrollo (embriología) y la medicina personalizada (Avsec y cols. Nature 2026). Mientras que la diana de AlphaMissense es el ADN codificante (2% del total), AlphaGenome analiza el 98% restante del ADN humano, que tiene funciones reguladoras (Lozano-Villalba & Puthanveettil. J Biol Chem 2026).
AlphaProteo es un programa de IA que permite diseñar nuevas proteínas capaces de unirse a moléculas diana, por ejemplo, antagonistas de receptores celulares de virus o inhibidores enzimáticos. De ese modo, puede acelerar el descubrimiento de nuevos fármacos (Zambaldi y cols. arXiv 2024). Por otro lado, surgen importantes dilemas éticos, entre otros: ¿Qué implicaciones puede tener la creación de proteínas que nunca han existido en la naturaleza? ¿Cómo garantizar que esta tecnología no se utilice con fines dañinos, como el diseño de patógenos artificiales?
Luces y sombras de las herramientas biotecnológicas de IA
Todos estos programas computacionales biotecnológicos usan técnicas avanzadas de deep learning y redes neuronales para resolver problemas de biología molecular que, de otro modo, requerirían experimentos muy difíciles o análisis computacionales muy complejos. La disponibilidad de estas herramientas biotecnológicas es un gran avance científico y está acelerando el conocimiento biomédico.
Hasta ahora, Google DeepMind no ha hecho pública la codificación y peso relativo de las variables seleccionadas en sus modelos bioinformáticos. En última instancia, solo un acceso abierto y completo a estas nuevas herramientas biotecnológicas puede ayudar a una mejor comprensión y desarrollo sin riesgos de aplicaciones biomédicas (García-González & Golewski. Trends Genet 2026).